Rozdział 9 Przykład badania rozkładu stopy zwrotu z akcji
9.1 Wprowadzenie
W tym opracowaniu będziemy analizować dzienne stopy zwrotu z akcji spółki PKOBP, która wchodzi w skład indeksu giełdowego WIG20. Indeks WIG20 to portfel akcji dwudziestu największych i najbardziej płynnych spółek notowanych na GPW w Warszawie. Jego skład jest ustalany po ostatniej sesji w styczniu (korekta roczna), kwietniu, lipcu i październiku (korekty kwartalne). Akutalny stan portfela WIG20 przedstawia poniższy wykres (rys. 9.1).
spolki1 <- c(15.00,13.06,11.44,8.95,8.24,8.17,7.17,3.98,3.94,3.00,2.40,
2.21,2.20,1.90,1.87,1.76,1.43,1.26,1.21,0.82)
val <- c('15.00%','13.06%','11.44%','8.95%','8.24%','8.17%','7.17%','3.98%','3.94%','3.00%',
'2.40%','2.21%','2.20%','1.90%','1.87%','1.76%','1.43%','1.26%','1.21%','0.82%')
nam <- c('PKOBP','PEKAO','KGHM','PZU','PGE','PKNORLEN','TPSA','TAURONPE','PGNIG',
'BZWBK','BRE','ASSECOPOL','GETIN','GTC','CEZ','TVN','PBG','POLIMEXMS',
'LOTOS','CYFRPLSAT')
par(mar=c(4,6,1,1)+0.1, mgp=c(3,0.6,0),las=1)
b <- barplot(spolki1, names.arg=nam, horiz=TRUE,xlim=c(0,18))
text(spolki1, b, labels=val, pos=4)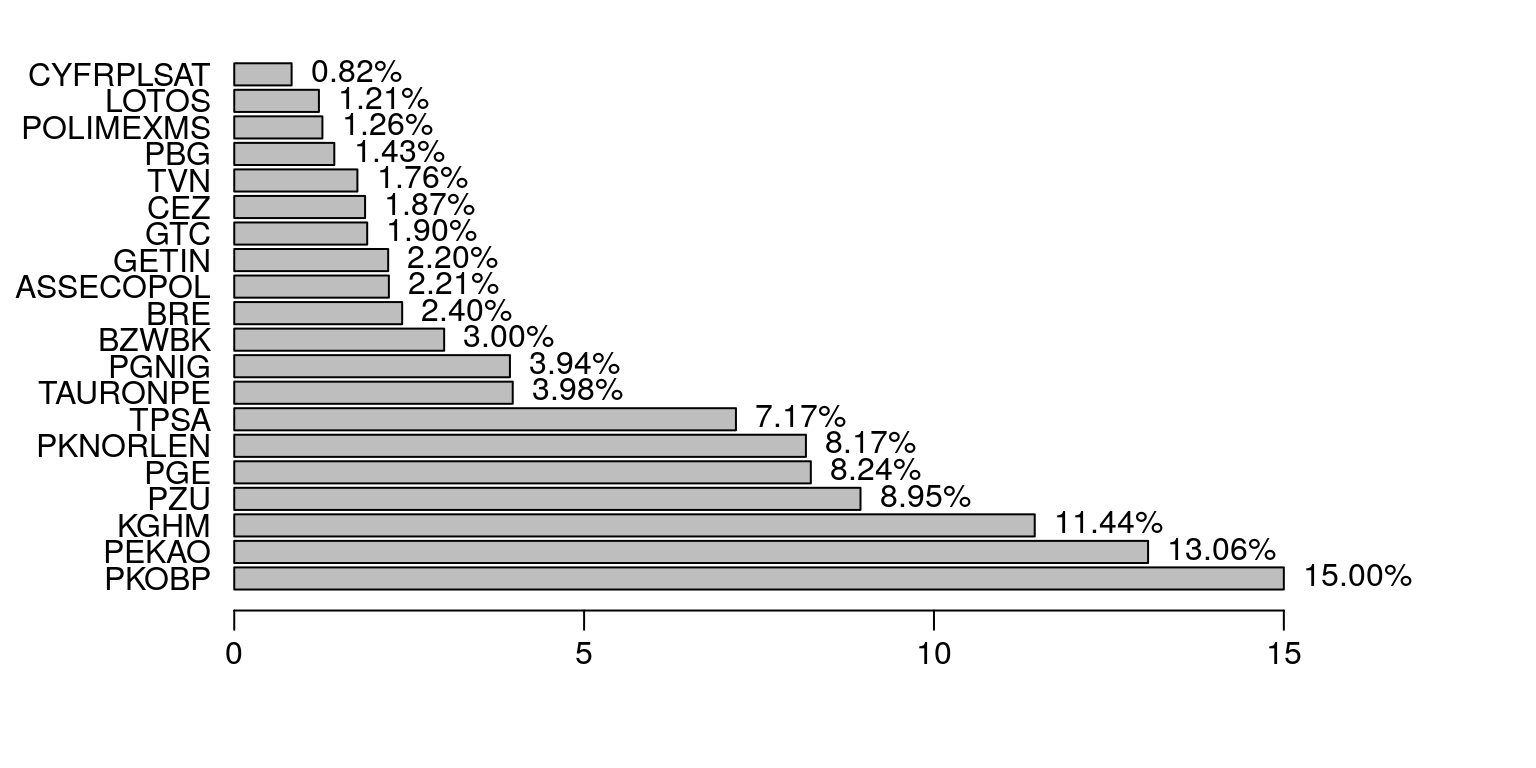
Rysunek 9.1: Struktura indeksu WIG20 – stan na 17 grudnia 2010 r.
9.2 Statystyki opisowe
Do naszych analiz zostanie wykorzystana stopa zwrotu spółki PKOBP, która ma największy udział w strukturze portfela WIG20 (rys. 9.1). Dane dotyczące notowań na warszawskiej giełdzie papierów wartościowych są dostępne w internecie. Przykładowo, po kliknięciu w odnośnik PKOBP zostaniemy przekierowani do serwisu internetowego z którego można pobrać interesujące nas dane. W kolejnym kroku można samodzielnie obliczyć logarytmiczne stopy zwrotu według wzoru: \[\begin{equation} z=\ln(Close)-\ln(Open)=\ln\left(\frac{Close}{Open}\right) \tag{9.1} \end{equation}\] gdzie: \(Close\) to cena zamknięcia oraz \(Open\) to cena otwarcia.
Innym rozwiązaniem jest pobranie już przetworzonego zestawu danych:
t <- read.csv("https://raw.githubusercontent.com/krzysiektr/datacsv/master/pkobp.csv")
head(t, 3)## Data Otwarcie Zamkniecie Maks Min Obrot_mln_zl z
## 1 2004-11-10 14.34 15.14 15.20 14.34 2780.40 0.054287413
## 2 2004-11-12 15.14 15.26 15.94 15.14 1049.56 0.007894778
## 3 2004-11-15 15.14 15.20 15.26 14.89 425.14 0.003955180par(mfcol=c(2,1),mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
plot(as.Date(t$Data),t$Zamkniecie,type="l",main="cena zamknięcia")
plot(as.Date(t$Data),t$z,type="l",main="stopa zwrotu")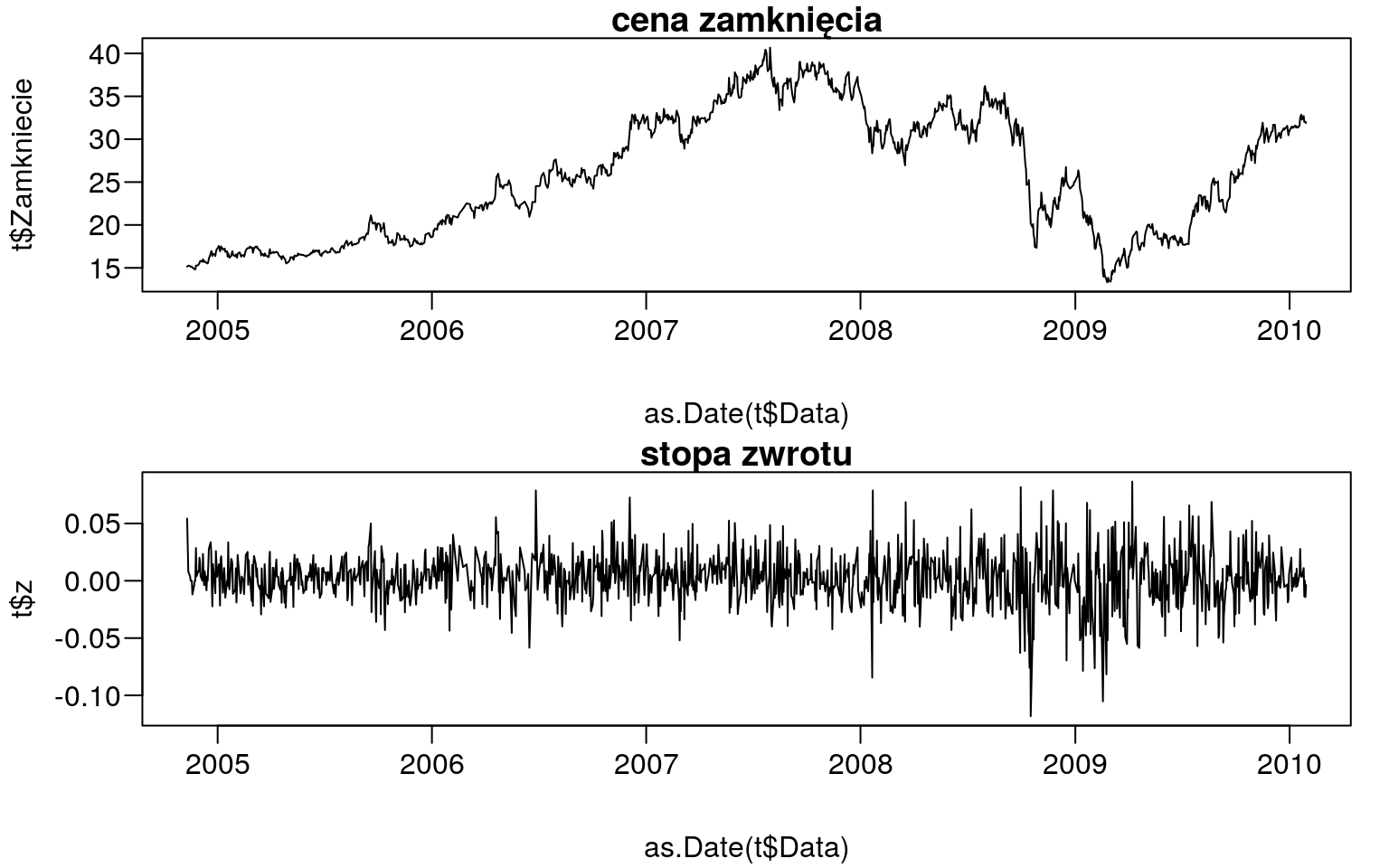
Rysunek 9.2: Stopy zwrotu i ceny zamknięcia od 10.11.2004 r. do 29.01.2010 r.
Poniżej obliczymy m.in. miary położenia tzn. średnią, medianę oraz kwartyle.
z <- t$z
summary(z)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -0.118224 -0.010002 0.001767 0.002169 0.014393 0.086565a także odchylenie standardowe według wzoru: \[\begin{equation} s=\sqrt{\frac{1}{n-1}\sum_{i=1}^{n}(x_i-\bar{x})^2} \tag{9.2} \end{equation}\]
sd(z)## [1] 0.02225315Z powyższych obliczeń wynika, że średnia stopa zwrotu w badanym okresie wyniosła \(0,22\%\), wartość maksymalna \(8,66\%\) a minimalna \(-11,82\%\). Natomiast w wypadku \(25\%\) sesji stopy zwrotu były mniejsze niż \(-0,01\%\), podczas \(50\%\) sesji wartości były mniejsze niż \(0,18\%\). Z kolei w przypadku \(75\%\) sesji zysk z akcji był mniejszy niż \(1,44\%\), a w przypadku pozostałych 25% sesji zysk był większy niż \(1,44\%\) (rys. 9.3).
Przy omawianiu różnych statystyk opisowych wato także wspomnieć o funkcji
quantile. Za jej pomocą mamy możliwość obliczenia wartość dowolnego kwantyla wykorzystując jedną z dziewięciu dostępnych metod. W
tym opracowaniu zaprezentujemy sposób obliczania kwantyli za pomocą metody
siódmej. W pierwszym kroku uporządkujemy nasz wektor danych w sposób rosnący:
sz <- sort(z) # wektor danych uporządkowany rosnącoNastępnie wyznaczymy numer kwantyla \(Q = 80\%\) według wzoru: \[\begin{equation} h=(n-1)\cdot Q+1 \tag{9.3} \end{equation}\]
h <- (length(z)-1)*0.8+1; h # numer kwantyla## [1] 1013.8Kolejnym krokiem będzie obliczenie wartości kwantyla na podstawie wzoru:
\[\begin{equation}
x_{\lfloor h \rfloor}+(h-{\lfloor h \rfloor})(x_{\lceil h\rceil}-x_{\lfloor h \rfloor})
\tag{9.4}
\end{equation}\]
gdzie: \(\lfloor h \rfloor\) to największa liczba całkowita nie większa od h oraz \(\lceil h \rceil\) to najmniejsza liczba całkowita nie mniejsza od h.
xL <- sz[floor(h)]; xP <- sz[ceiling(h)]
xL+(h-floor(h))*(xP-xL) # wartość kwantyla## [1] 0.01779921Teraz wykorzystamy gotową funkcję z pakietu stats:
quantile(z,0.80,type=7)## 80%
## 0.01779921Otrzymane wartości kwantyli możemy również zaznaczyć na wykresie (rys. 9.3).
q <- as.numeric(quantile(z,c(0.25,0.5,0.75,0.80)))
par(mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
plot(ecdf(z))
arrows(q,c(0.25,0.5,0.75,0.80),q,c(0,0,0,0),length=0.1,angle=15,
col=c("violetred3","YellowGreen","SteelBlue","wheat4"))
arrows(q,c(0.25,0.5,0.75,0.80),c(-5,-5,-5,-5),c(0.25,0.5,0.75,0.8),angle=0,
col=c("violetred3","YellowGreen","SteelBlue","wheat4"))
points(q,c(0.25,0.5,0.75,0.8),bg="white",pch=21,
col=c("violetred3","YellowGreen","SteelBlue","wheat4"))
legend("right", paste(c("Q25=", "Q50=", "Q75=", "Q80="),round(q,4)),bty="n",#lty=1,
text.col=c("violetred3","YellowGreen","SteelBlue","wheat4"))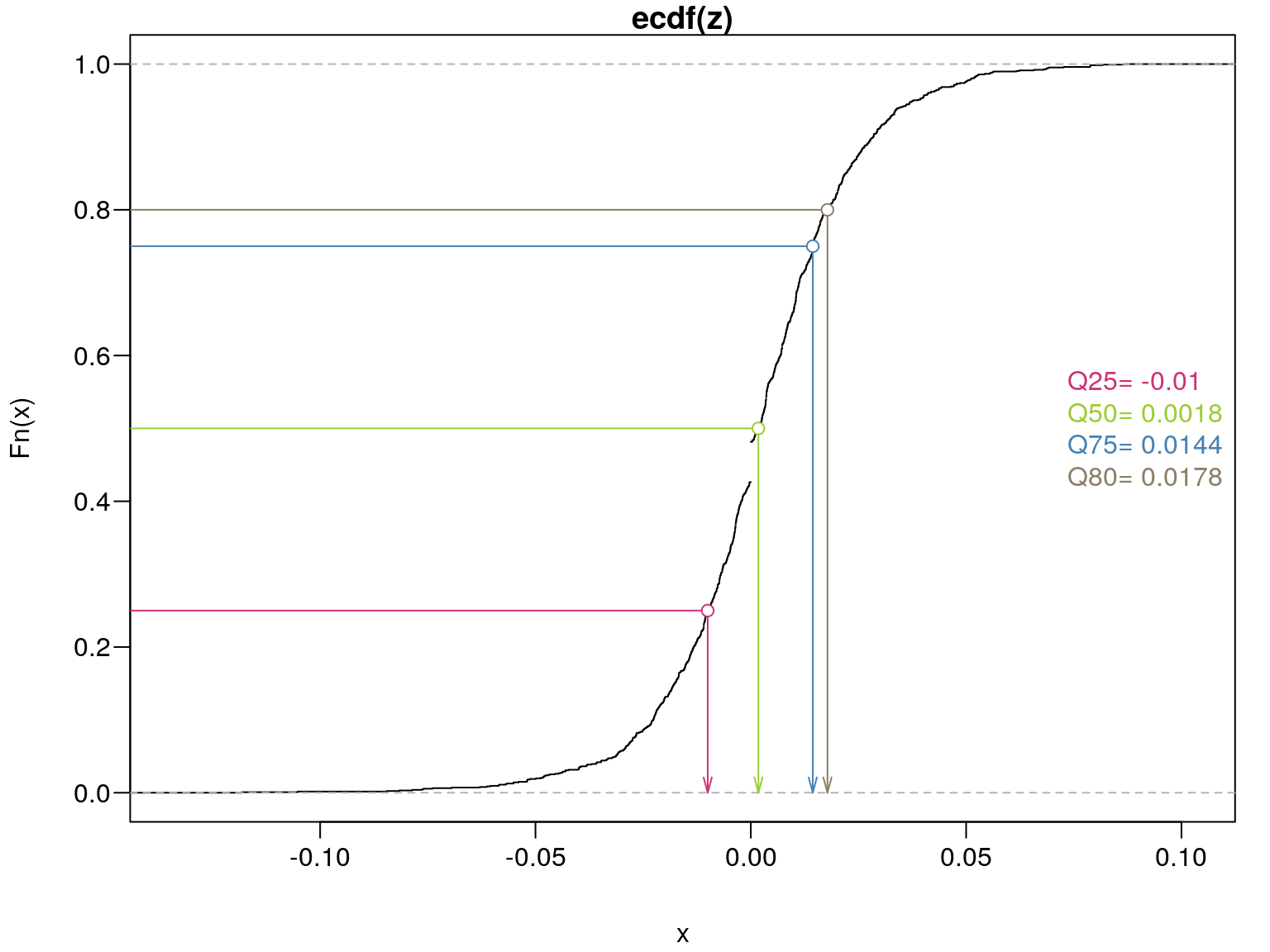
Rysunek 9.3: Dystrybuanta – prezentacja graficzna kwantyli.
Wyznaczmy jeszcze wzory dla pozostałych metod szcowania kwantyli. Dla me- tod od 4 do 9 algorytmy różnią się tylko sposobem wyznaczenia numeru kwantyla. Wartość kwantyla jest obliczana tak jak to zostało przedstawione w powyższym przykładzie czyli według wzoru (9.4)
- typ 4:
\[\begin{equation} h=n\cdot Q \tag{9.5} \end{equation}\]
- typ 5:
\[\begin{equation} h=n\cdot Q + 1/2 \tag{9.6} \end{equation}\]
- typ 6:
\[\begin{equation} h=(n+1)\cdot Q \tag{9.7} \end{equation}\]
- typ 7:
\[\begin{equation} h=(n-1)\cdot Q + 1 \tag{9.8} \end{equation}\]
- typ 8:
\[\begin{equation} h=(n+1/3)\cdot Q + 1/3 \tag{9.9} \end{equation}\]
- typ 9:
\[\begin{equation} h=(n+1/4)\cdot Q + 3/8 \tag{9.10} \end{equation}\]
W przypadku metod od 1 do 3 do wyznaczenia numeru kwantyla oraz jego wartości korzystamy z nastęujących wzorów:
- typ 1:
\[\begin{equation} h=n\cdot Q +1/2 \quad\wedge\quad x_{\lceil h-1/2\rceil} \tag{9.11} \end{equation}\]
- typ 2:
\[\begin{equation} h=n\cdot Q +1/2 \quad\wedge\quad (x_{\lceil h-1/2\rceil}+x_{\lfloor h+1/2\rfloor})/2 \tag{9.12} \end{equation}\]
- typ 3:
\[\begin{equation} h=n\cdot Q \quad\wedge\quad x_{[h]} \tag{9.13} \end{equation}\]
Za pomocą komendy quantile można także wyznaczyć jednocześnie kilka wartości
dowolnych kwantyli:
quantile(z,c(0.17,0.60,0.83),type=7)## 17% 60% 83%
## -0.015450594 0.006886427 0.020418864W różnych opracowaniach często spotykamy się z następującymi wzorami dotyczącymi skośności: \[\begin{equation} S_{1}=\frac{\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^3}{\sqrt{\left(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^2\right)^3}} \tag{9.14} \end{equation}\]
(S1 <- e1071::skewness(z,type=1))## [1] -0.2199644\[\begin{equation} S_{2}=\frac{\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^3\frac{\sqrt{n(n-1)}}{n-2}}{\sqrt{\left(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^2\right)^3}} \tag{9.15} \end{equation}\]
(S2 <- e1071::skewness(z,type=2))## [1] -0.2202252\[\begin{equation} S_{3}=\frac{\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^3}{s^3} \tag{9.16} \end{equation}\] gdzie \(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^3\) oraz \(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^2\) to odpowiednio trzeci i drugi moment centralny oraz s to odchylenie standardowe z próby.
(S3 <- e1071::skewness(z,type=3))## [1] -0.219704Ponieważ obliczony wskaźnik skośności jest równy \(-0,22\) możemy sądzić, że rozkład stopy zwrotu charakteryzuje się niewielką lewostronną skośnością.
moments::agostino.test(z)##
## D'Agostino skewness test
##
## data: z
## skew = -0.21996, z = -3.17810, p-value = 0.001483
## alternative hypothesis: data have a skewnessRównież w przypadku miar koncentracji możemy spotkać się kilkoma wzorami na kurtozę: \[\begin{equation} K_{1}=\frac{\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^4}{\left(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^2\right)}-3 \tag{9.17} \end{equation}\]
(K1 <- e1071::kurtosis(z,type=1))## [1] 2.315957\[\begin{equation} K_{2}=\frac{n(n+1)}{(n-1)(n-2)(n-3)}\sum_{i=1}^{n}\left(\frac{x_i-\bar{x}}{s}\right)^4-\frac{3(n-1)^2}{(n-2)(n-3)} \tag{9.18} \end{equation}\]
(K2 <- e1071::kurtosis(z,type=2))## [1] 2.329873\[\begin{equation} K_{3}=\frac{\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^4}{s^4}-3 \tag{9.19} \end{equation}\] gdzie \(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^4\) oraz \(\frac{1}{n}\sum_{i=1}^{n}(x_i-\bar{x})^2\) to odpowiednio czwarty i drugi moment centralny oraz \(s\) to odchylenie standardowe z próby.
(K3 <- e1071::kurtosis(z,type=3))## [1] 2.307569Wysoka wartość kurtozy świadczy o tym, że rozkład stóp zwrotu charakteryzuje się spiczastością (rozkład leptokurtyczny) w stosunku do rozkładu normalnego (rozkład mezokurtyczny).
moments::anscombe.test(z)##
## Anscombe-Glynn kurtosis test
##
## data: z
## kurt = 5.3160, z = 8.5299, p-value < 2.2e-16
## alternative hypothesis: kurtosis is not equal to 39.3 Rozkład normalny
Funkcja gęstości rozkładu normalnego \(N(\mu,\sigma)\) dla \(\mu\in R\) oraz \(\sigma>0\) czyli dnorm jest przedstawiona poniżej:
\[\begin{equation}
f(x)=\frac{1}{\sigma\sqrt(2\pi)}\exp\left(\frac{-(x-\mu)^2}{2\sigma^2}\right)
\tag{9.20}
\end{equation}\]
gdzie: \(\mu\) to średnia, natomiast \(\sigma\) to odchylenie standardowe.
Do estymacji nieznanych parametrów rozkładu normalnego wykorzystamy następujące funkcję:
a <- mean(z); a # średnia## [1] 0.00216913b <- sd(z); b # odchylenie standardowe## [1] 0.02225315W przypadku gdybyśmy chcieli estymować parametry \(\mu\) oraz \(\sigma\) za pomocą funkcji nlminb kod wyglądałby następująco:
# logarytm funkcji wiarygodności:
f1 <- function(theta, z) {
sum(-dnorm(z, mean=theta[1], sd=theta[2], log=TRUE))
}
# parametry startowe:
p.start <- c(mean(z), sd(z))
# optymalizacja funkcji f1:
e1 <- nlminb(p.start, f1, z=z, lower=c(-Inf,0), upper=c(Inf,Inf))
e1[c('par','objective')]## $par
## [1] 0.002169126 0.022244371
##
## $objective
## [1] -3023.984Jak można zauważyć otrzymane parametry są dokładnie takie same jak te, które
oszacowaliśmy za pomocą funkcji mean i sd.
W środowisku R można oszacować nieznane parametry również za pomocą funkcji MASS::fitdistr lub bardziej rozbudowanej wersji fitdistrplus::fitdist. Dla wbudowanych dystrybuant nie ma konieczności podawania parametrów startowych oraz ograniczeń przedziałowych. Takie opcje są bardzo użyteczne jeśli chcemy szukać parametrów dla dystrybuant zdefiniowanych przez nas.
summary(fitdistrplus::fitdist(z,'norm'))## Fitting of the distribution ' norm ' by maximum likelihood
## Parameters :
## estimate Std. Error
## mean 0.00216913 0.0006249307
## sd 0.02224437 0.0004378806
## Loglikelihood: 3023.984 AIC: -6043.968 BIC: -6033.679
## Correlation matrix:
## mean sd
## mean 1 0
## sd 0 1Znając już parametry \(\mu\) oraz \(\sigma\) możemy teraz przeprowadzić test zgodności Andersona-Darlinga i odpowiedzieć na pytanie: czy stopa zwrotu PKOBP pochodzi z rozkładu normalnego?
ADGofTest::ad.test(z,pnorm,mean(z),sd(z))##
## Anderson-Darling GoF Test
##
## data: z and pnorm
## AD = 7.2474, p-value = 0.0002546
## alternative hypothesis: NAPonieważ \(p-value = 0.0002546\) to hipotetę zerową należy odrzucić. Zatem na poziomie istotności \(\alpha = 0,05\) należy stwierdzić, że stopa zwrotu PKOBP nie ma rozkładu normalnego. Naszą decyzję potwierdza także (rys. 9.4).
par(mfcol=c(1,2),mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
plot(density(z),col='SteelBlue',lwd=4,main='gęstość')
curve(dnorm(x,mean(z),sd(z)),add=TRUE,col='violetred3',lwd=3)
legend("topright",bg='white',bty="n",cex=1,lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))
plot(ecdf(z),col='SteelBlue',lwd=4,main='dystrybuanta')
curve(pnorm(x,mean(z),sd(z)),add=TRUE,col='violetred3',lwd=3)
legend("topleft",bg='white',bty="n",lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))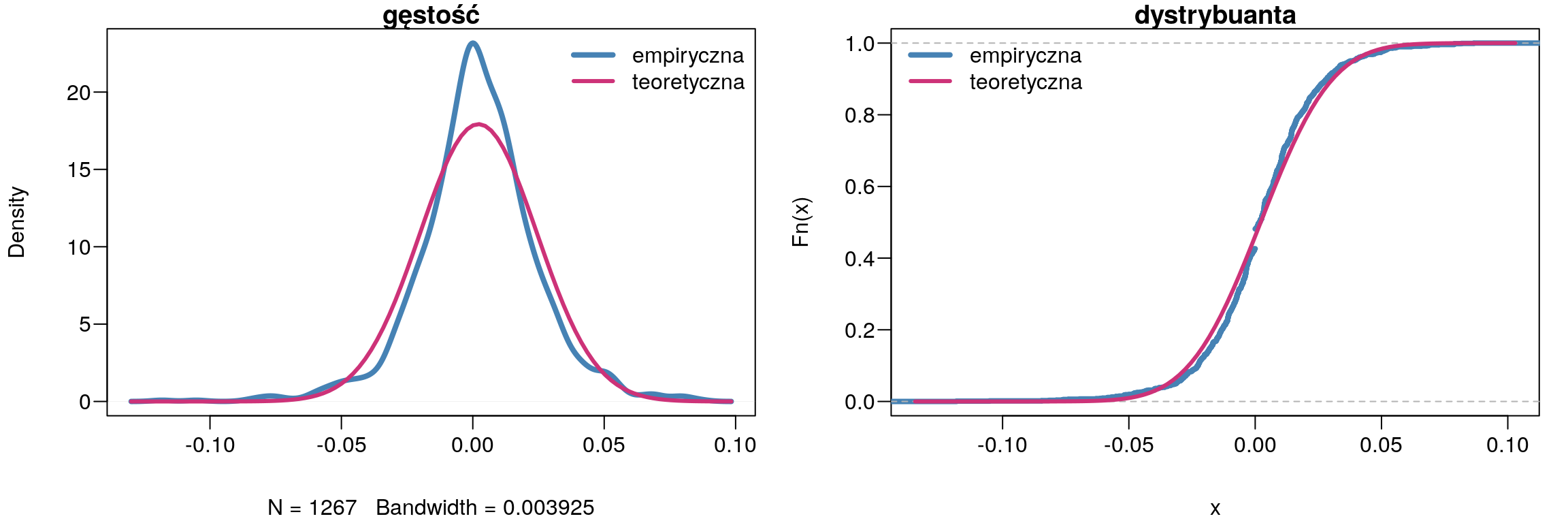
Rysunek 9.4: Porównanie wykresu gęstości z rozkładem normalnym.
9.4 Rozkład Cauchy’ego
Funkcja gęstości rozkładu Cauchy’ego \(C(a, b)\) dla \(a \in R\) oraz \(b > 0\) czyli dcauchy jest przedstawiona poniżej:
\[\begin{equation}
f(x)=\frac{1}{\pi b \left(1+\left(\frac{x-a}{b}\right)^2\right)}
\tag{9.21}
\end{equation}\]
gdzie: \(a\) to parametr położenia, natomiast \(b\) to parametr skali.
Tak samo jak w przypadku rozkładu normalnego, aby wykorzystać funkcję nlminb należy oszacować parametry startowe. Dla rozkładu Cauchy’ego można to zrobić w
następujący sposób:
a <- median(z); a # parametr położenia: a## [1] 0.001766785b <- IQR(z)/2; b # parametr skali: b## [1] 0.0121972Teraz metodą największej wiarygodności oszacujemy parametry rozkładu Cauche’go.
# logarytm funkcji wiarygodności:
f2 <- function(theta, z) {
sum(-dcauchy(z, location=theta[1], scale=theta[2], log=TRUE))
}
# parametry startowe:
p.start <- c(median(z), IQR(z)/2)
# optymalizacja funkcji f2:
e2 <- nlminb(p.start, f2, z=z, lower=c(-Inf,0), upper=c(Inf,Inf))
e2[c('par','objective')]## $par
## [1] 0.002025263 0.011231456
##
## $objective
## [1] -2945.315Prawie identyczne wyniki otrzymamy przy wykorzystaniu funkcji fitdistrplus::fitdist.
summary(fitdistrplus::fitdist(z,'cauchy'))## Fitting of the distribution ' cauchy ' by maximum likelihood
## Parameters :
## estimate Std. Error
## location 0.002029574 0.0004912212
## scale 0.011230842 0.0004071368
## Loglikelihood: 2945.315 AIC: -5886.63 BIC: -5876.341
## Correlation matrix:
## location scale
## location 1.00000000 0.03089075
## scale 0.03089075 1.00000000Do sprawdzenia zgoności rozkładu empirycznego z rozkładem Cauchy’ego zastosujemy test Andersona-Darlinga.
ADGofTest::ad.test(z,pcauchy, 0.002029574, 0.011230842)##
## Anderson-Darling GoF Test
##
## data: z and pcauchy
## AD = 10.36, p-value = 3.782e-06
## alternative hypothesis: NATakże i w tym razem na poziomie istotności \(\alpha = 0,05\) odrzucamy hipotezę zerową, która zakłada, że stopa zwrotu ma rozkład Cauchy’ego.
par(mfcol=c(1,2),mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
plot(density(z),col='SteelBlue',lwd=4,main='gęstość',ylim=c(0,30))
curve(dcauchy(x,0.002029574, 0.011230842),add=TRUE,col='violetred3',lwd=3)
legend("topright",bg='white',bty="n",cex=1,lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))
plot(ecdf(z),col='SteelBlue',lwd=4,main='dystrybuanta')
curve(pcauchy(x,0.002029574, 0.011230842),add=TRUE,col='violetred3',lwd=3)
legend("topleft",bg='white',bty="n",lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))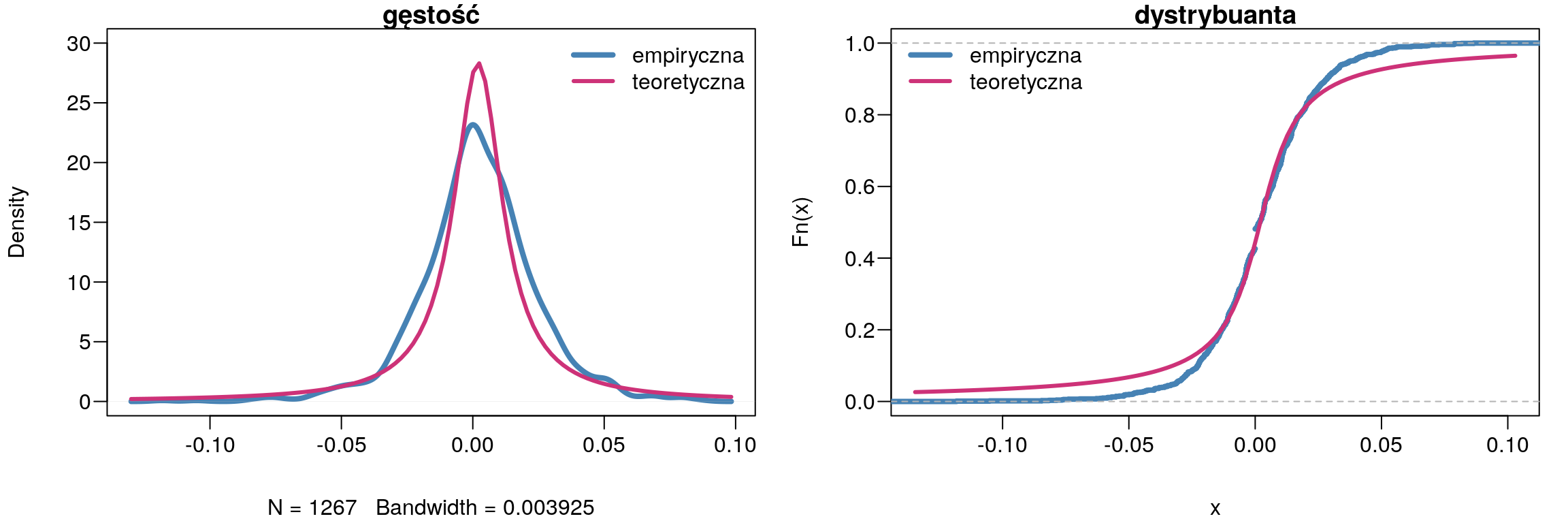
Rysunek 9.5: Porównanie wykresu gęstości z rozkładem Cauchy`ego.
9.5 Rozkład Laplace’a
Funkcja gęstości rozkładu Laplace’a \(L(a, b)\) dla \(a \in R\) oraz \(b > 0\) czyli VGAM::dlaplace jest przedstawiona poniżej:
\[\begin{equation}
f(x)=\frac{1}{2b}\exp\left(\frac{-|x-a|}{b}\right)
\tag{9.22}
\end{equation}\]
gdzie: \(a\) to parametr położenia, natomiast \(b\) to parametr skali.
Parametry startowe oszacujemy w następujący sposób:
a <- median(z); a # parametr położenia: a## [1] 0.001766785b <- mean(abs(z-median(z))); b # parametr skali: b## [1] 0.01628686Mając do dyspozycji funkcję gęstości VGAM::dlaplace oraz parametry startowe dokonamy estymacji parametrów a oraz b dla rozkładu Laplace’a.
# logarytm funkcji wiarygodności:
f3 <- function(theta, z) {
sum(-VGAM::dlaplace(z, location=theta[1], scale=theta[2], log=TRUE))
}
# parametry startowe:
p.start <- c(median(z), mean(abs(z-median(z))))
# optymalizacja funkcji f3:
e3 <- nlminb(p.start, f3, z=z, lower=c(-Inf,0), upper=c(Inf,Inf))
e3## $par
## [1] 0.001766785 0.016286861
##
## $objective
## [1] -3071.524
##
## $convergence
## [1] 1
##
## $iterations
## [1] 1
##
## $evaluations
## function gradient
## 25 2
##
## $message
## [1] "false convergence (8)"Stosowanie algorytmów optymalizacyjnych ogólnego przeznaczenia np. nlminb do wyznaczania parametrów funkcji
wymaga podawania parametrów startowych. Jeśli nie chcemy ich podawać
warto skorzystać z funkcj VGAM::vglm.
Za jej pomocą można oszacować szukane parametry dla wielu ciekawych rozkładów np. Laplace’a.
fit <- VGAM::vglm(z ~ 1, VGAM::laplace, data.frame(z), trace=FALSE, crit="l")
VGAM::Coef(fit)## location scale
## 0.001766785 0.016296640ADGofTest::ad.test(z,VGAM::plaplace, 0.001766785, 0.016296640)##
## Anderson-Darling GoF Test
##
## data: z and VGAM::plaplace
## AD = 1.4474, p-value = 0.1896
## alternative hypothesis: NAPonieważ \(p-value = 0.1896\), więc nie ma podstaw do odrzucenia hipotezy zerowej. Zatem można przyjąć, że rozkład stóp zwrotu spółki PKOBP ma rozkład Laplace’a.
par(mfcol=c(1,2),mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
plot(density(z),col='SteelBlue',lwd=4,main='gęstość',ylim=c(0,30))
curve(VGAM::dlaplace(x,0.001766785, 0.016296640),add=TRUE,col='violetred3',lwd=3)
legend("topright",bg='white',bty="n",cex=1,lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))
plot(ecdf(z),col='SteelBlue',lwd=4,main='dystrybuanta')
curve(VGAM::plaplace(x,0.001766785, 0.016296640),add=TRUE,col='violetred3',lwd=3)
legend("topleft",bg='white',bty="n",lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))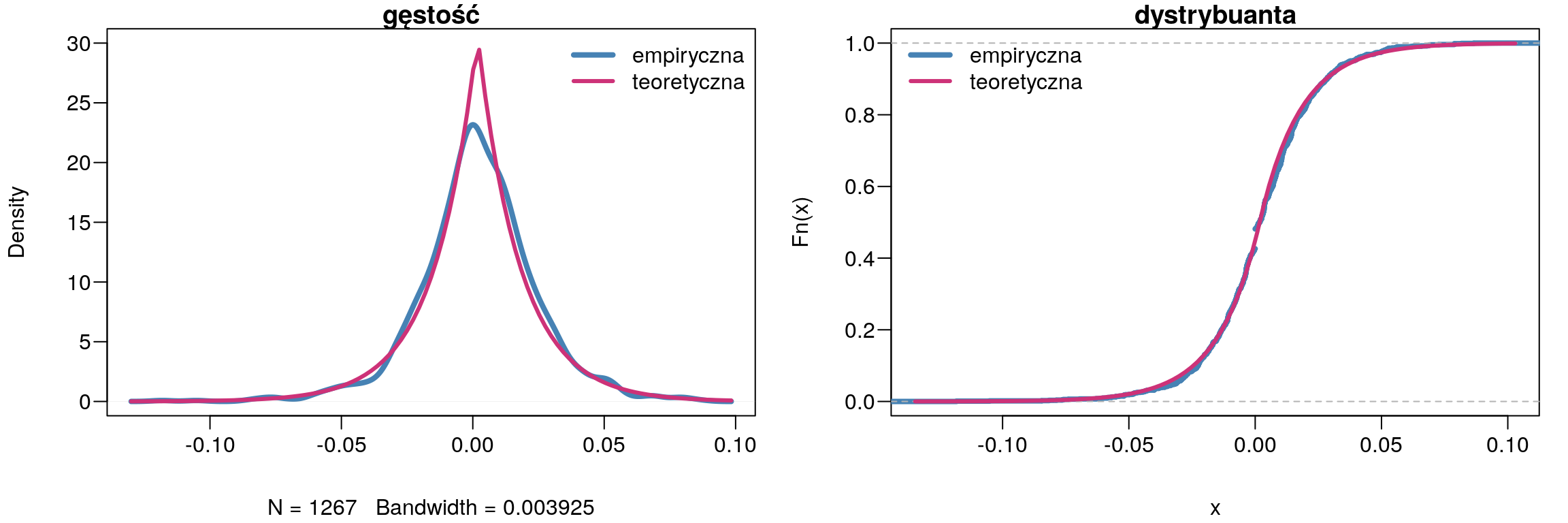
Rysunek 9.6: Porównanie wykresu gęstości z rozkładem Laplace`a.
9.6 Rozkład Stabilny
Rozkład stabilny \(S(\alpha, \beta, \gamma, \delta)\), gdzie \(\alpha \in(0; 2)\) to parametr kształtu, \(\beta \in(-1; 1)\)
to indeks skośności, \(\gamma > 0\) to parametr skali, oraz \(\delta \in R\) to parametr położenia,
nie ma ściśle określonej funkcji gęstości – stabledist::dstable. W zależności od wartości parametrów \(\alpha\)
oraz \(\beta\) możemy otrzymać rozkłady, takie jak: rozkład normalny: \(\alpha = 2\), rozkład
Cauchy’ego: \(\alpha = 1\) i \(\beta = 0\) oraz rozkład Levy’ego: \(\alpha = 12\) i \(\beta = -1\).
Do oszacowania parametrów rozkładu stabilnego można wykorzystać funkcję libstableR::stable_fit_mle która stosuje metodę największej wiarygodności.
p <- libstableR::stable_fit_mle(z)## INIT MCCULLCOHp## [1] 1.7281736156 -0.0007958884 0.0132426670 0.0023446287ADGofTest::ad.test(z,stabledist::pstable,p[1],p[2],p[3],p[4])##
## Anderson-Darling GoF Test
##
## data: z and stabledist::pstable
## AD = 0.72666, p-value = 0.5369
## alternative hypothesis: NATest Andersona-Darlinga wykazał, że badana zmienna ma rozkład stabilny o parametrach: \(\alpha = 1,73\), \(\beta = -0,000796\), \(\gamma = 0,01\) oraz \(\delta = 0,002\). Warto zwrócić uwagę na wysokość p-value dla rozkładu stabilnego, które wynosi \(0,5369\) i jest większa niż dla rozkładu Laplace’a \((0,1896)\). A zatem należy sądzić, że właśnie ten rozkład lepiej opisuje badaną zmienną (rys. 9.7).
par(mfcol=c(1,2),mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
plot(density(z),col='SteelBlue',lwd=4,main='gęstość')
curve(stabledist::dstable(x, 1.7281736156, -0.0007958884, 0.0132426670, 0.0023446287),
add=TRUE,col='violetred3',lwd=3)
legend("topright",bg='white',bty="n",cex=1,lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))
plot(ecdf(z),col='SteelBlue',lwd=4,main='dystrybuanta')
curve(stabledist::pstable(x, 1.7281736156, -0.0007958884, 0.0132426670, 0.0023446287),
add=TRUE,col='violetred3',lwd=3)
legend("topleft",bg='white',bty="n",lty=1,lwd=c(4,3),
c('empiryczna','teoretyczna'),col=c('SteelBlue','violetred3'))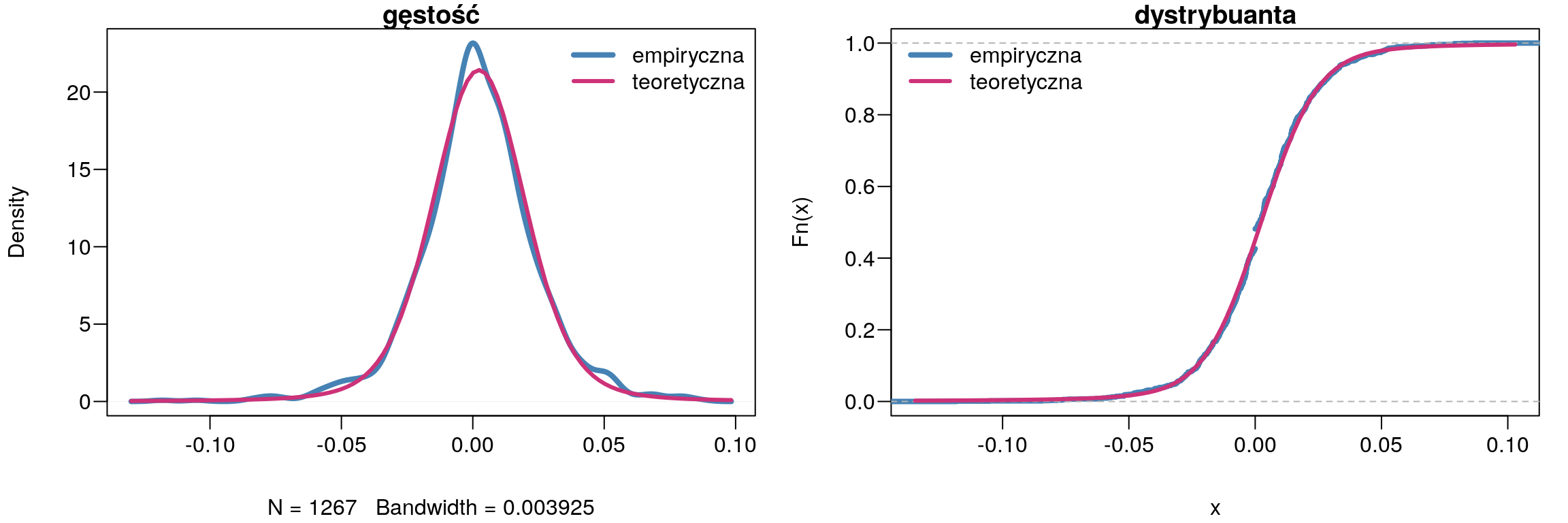
Rysunek 9.7: Porównanie wykresu gęstości z rozkładem Stabilnym.
Znając rozkład badanej zmiennej można także obliczyć prawdopodobieństwo wystąpienia określonej stopy zwrotu. W tym celu wykorzystamy funkcję stabledist::pstable
czyli dystrybuantę rozkładu stabilnego.
- Jakie jest prawdopodobieństwo, że stopa zwrotu będzie mniejsza niż \(2\%\)? \[P(z<0,02)\]
a <- p[1]
b <- p[2]
g <- p[3]
d <- p[4]
stabledist::pstable(0.02, a,b,g,d)## [1] 0.821845par(mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
col='white', main='prawdopodobieństwo')
x <- seq(0.02,-0.1,length=300)
y <- stabledist::dstable(x,a,b,g,d)
polygon(c(0.02,x,-0.1),c(0,y,0),
col='Violet',border = "Dark Magenta")
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
lwd=4,col='Dark Magenta',add=TRUE)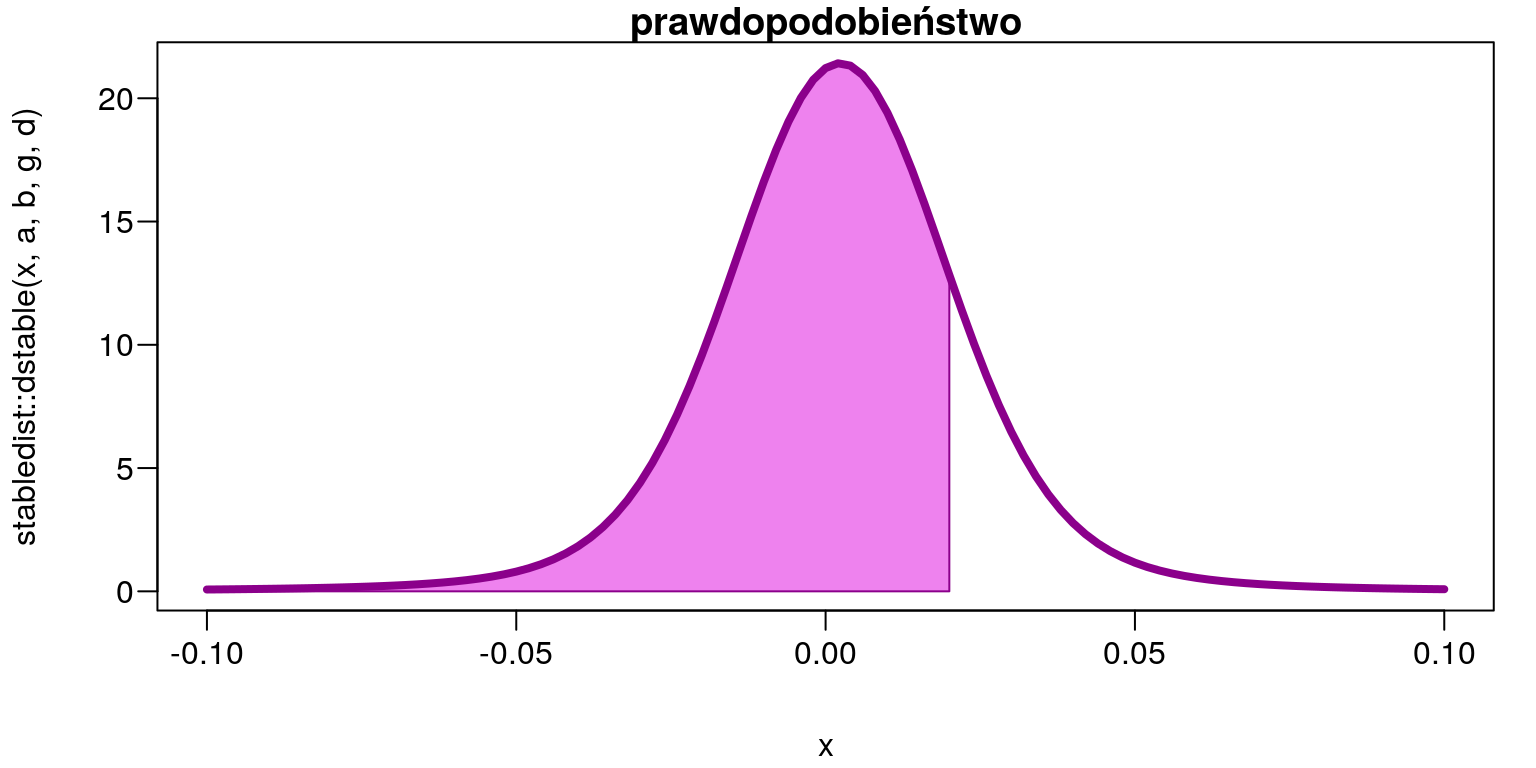
Rysunek 9.8: Obszar prawdopodobieństwa \(P(z<0,02)=0,821845\)
- Jakie jest prawdopodobieństwo, że stopa zwrotu będzie większa niż \(3,2\%\)? \[P(z > 0,032)\]
1-stabledist::pstable(0.032, a,b,g,d)## [1] 0.07146513par(mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
col='white',main='prawdopodobieństwo')
x <- seq(0.032,0.1,length=300)
y <- stabledist::dstable(x,a,b,g,d)
polygon(c(0.032,x,0.1),c(0,y,0),
col='Violet',border = "Dark Magenta")
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
lwd=4,col='Dark Magenta',add=TRUE)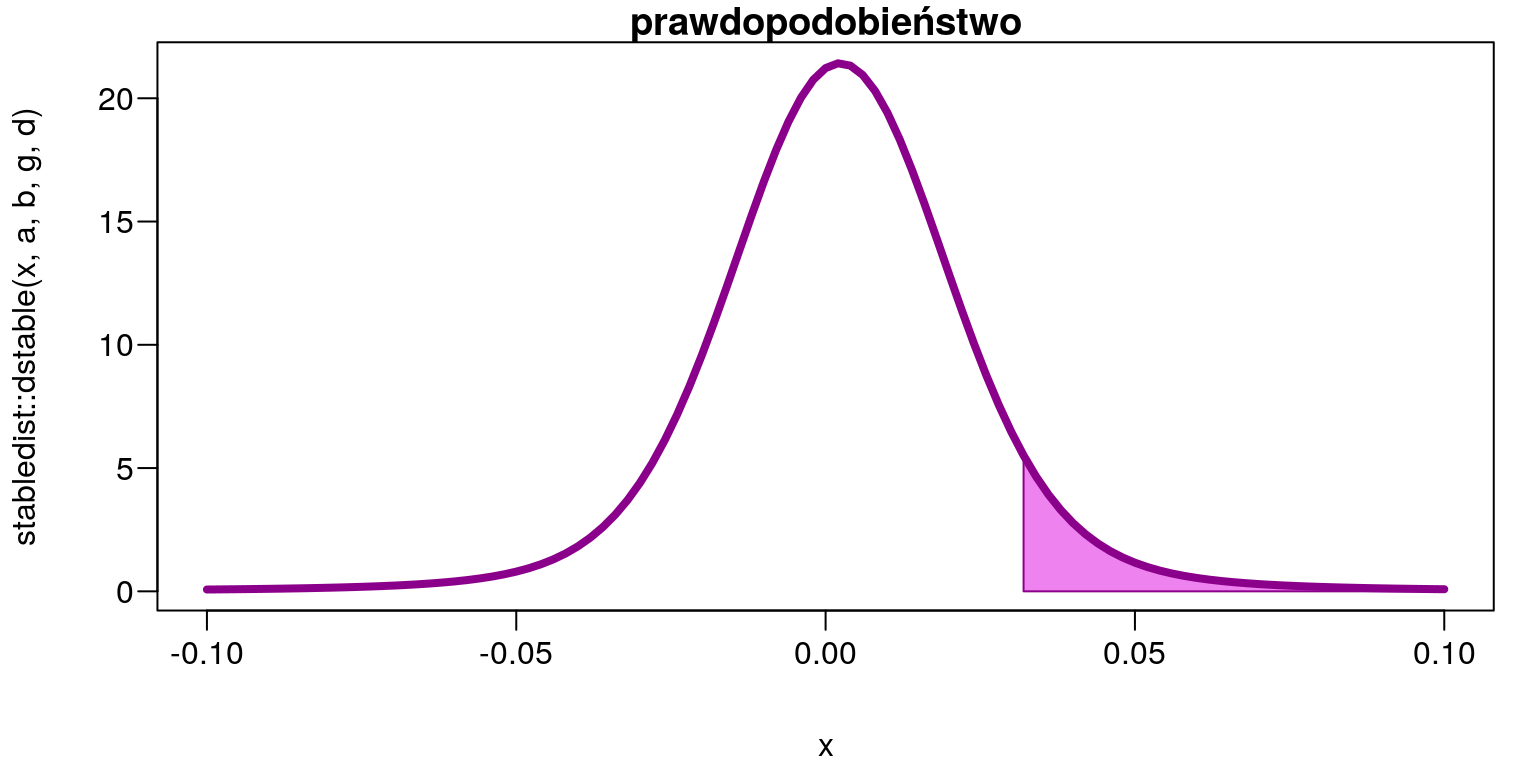
Rysunek 9.9: Obszar prawdopodobieństwa \(P(z>0,032)=0,07146513\)
- Jakie jest prawdopodobieństwo, że stopa zwrotu będzie się zawierać w przedziale \((0\%; 3,78\%)\)? \[P(0<z<0,0378)\]
stabledist::pstable(0.0378, a,b,g,d)-stabledist::pstable(0, a,b,g,d)## [1] 0.5038598par(mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
col='white',main='prawdopodobieństwo')
x <- seq(0,0.0378,length=300)
y <- stabledist::dstable(x,a,b,g,d)
polygon(c(0,x,0.0378),c(0,y,0),
col='Violet',border = "Dark Magenta")
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
lwd=4,col='Dark Magenta',add=TRUE)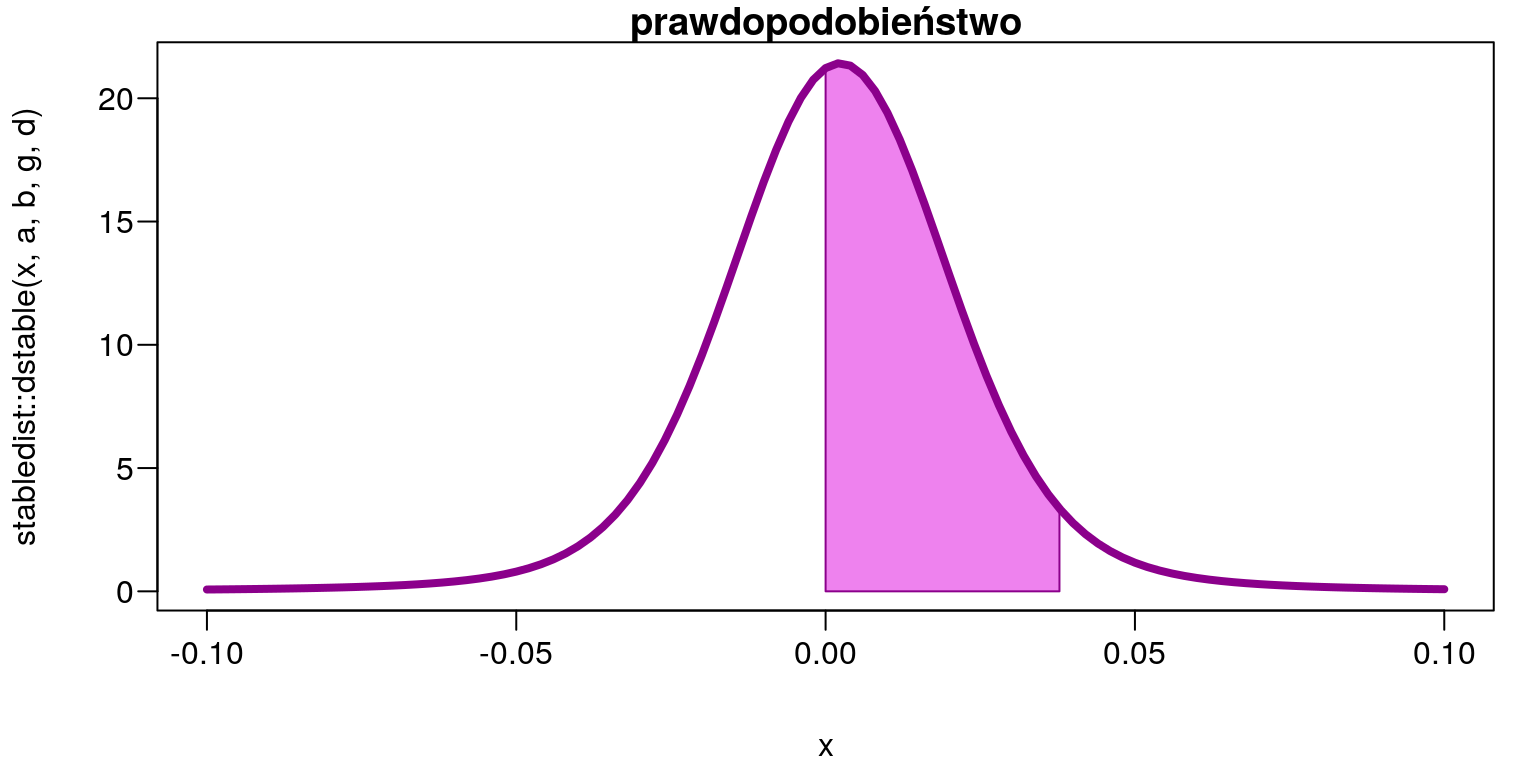
Rysunek 9.10: Obszar prawdopodobieństwa \(P(0<z<0,0378)=0,5038598\)
- Jakie jest prawdopodobieństwo, że stopa zwrotu będzie mniejsza niż \(1,75\%\) lub większa niż \(3,78\%\)? \[P (0,0175 > z > 0,0378)\]
stabledist::pstable(0.0175, a,b,g,d)+(1-stabledist::pstable(0.0378, a,b,g,d))## [1] 0.8336819par(mar=c(4,4,1,1)+0.1, mgp=c(3,0.6,0),las=1)
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
col='white',main='prawdopodobieństwo')
x <- seq(-0.1,0.0175,length=300)
y <- stabledist::dstable(x,a,b,g,d)
polygon(c(-0.1,x,0.0175),c(0,y,0),
col='Violet',border = "Dark Magenta")
x <- seq(0.0378,0.1,length=300)
y <- stabledist::dstable(x,a,b,g,d)
polygon(c(0.0378,x,0.1),c(0,y,0),
col='Violet',border = "Dark Magenta")
curve(stabledist::dstable(x,a,b,g,d),-0.1,0.1,
lwd=4,col='Dark Magenta',add=TRUE)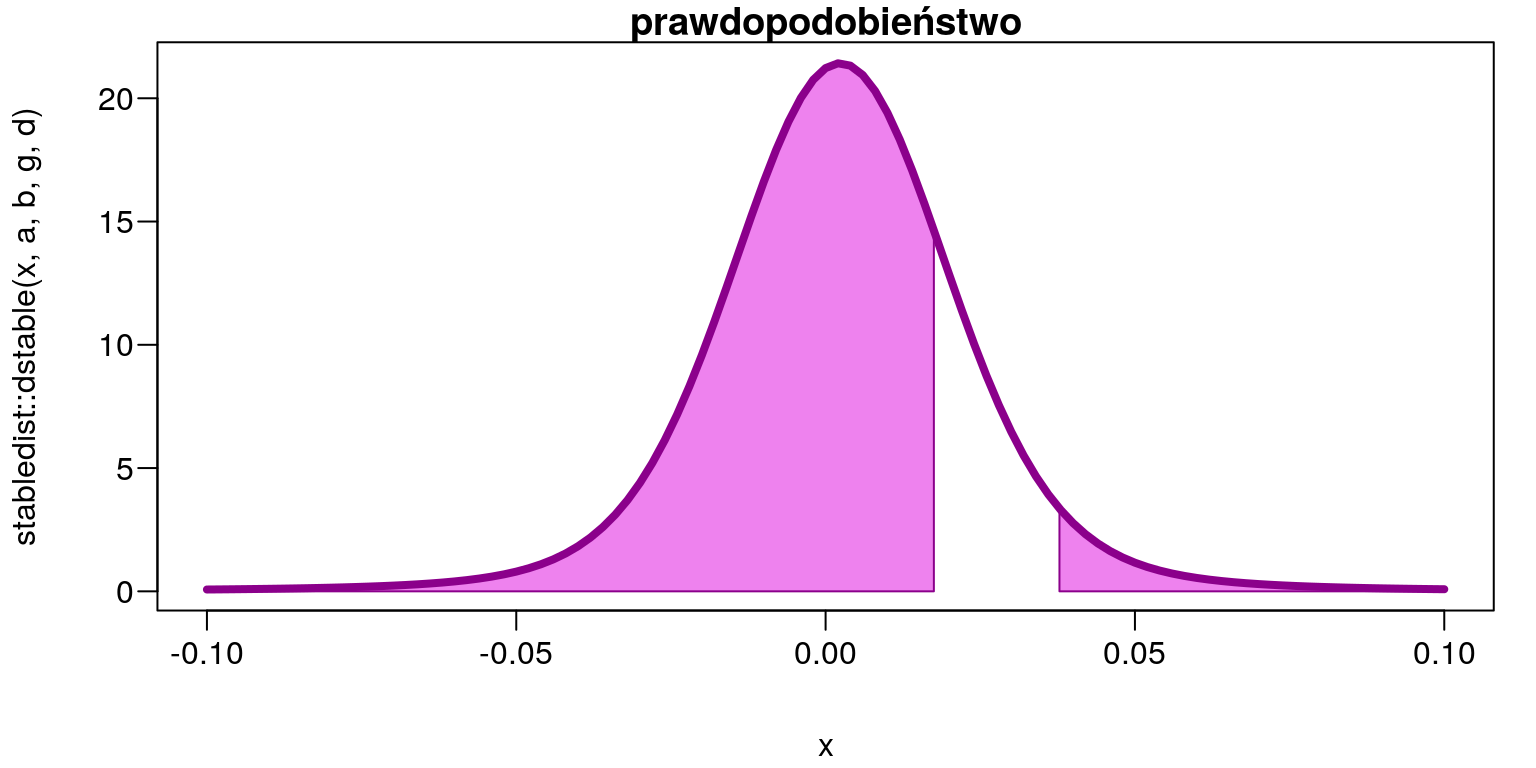
Rysunek 9.11: Obszar prawdopodobieństwa \(P(0,0175>z>0,0378)=0,8336819\)
Na zakończenie warto jeszcze wspomnieć, że środowisko R oferuje także wiele innych rozkładów prawdopodobieństwa wraz z funkcjami do szacowania
ich parametrów. W paczce fBasics oprócz funkcji do estymacji parametrów rozkładu stabilnego fBasic::stableFit mamy do dyspozycji takie rozkłady jak:
odwrotny rozkład normalny (Normal Inverse Gaussian Distribution) gdzie:
dnig– funkcja gęstości,pnig– dystrybuanta,qnig– kwantyle,rnig– zmienne losowe,nigFit– estymacja parametrów.rozkład hiperboliczny (Hyperbolic Distribution) gdzie:
dhyp– funkcja gęstości,phyp– dystrybuanta,qhyp– kwantyle,rhyp– zmienne losowe,hypFit– estymacja parametrów.uogolniony rozkład hiperboliczy (Generalized Hyperbolic Distribution) gdzie:
dgh– funkcja gęstości,pgh– dystrybuanta,qgh– kwantyle,rgh– zmienne losowe,ghFit– estymacja parametrów.uogolniony rozkład hiperboliczy t-Studenta (Generalized Hyperbolic Student-t) gdzie:
dght– funkcja gęstości,pght– dystrybuanta,qght– kwantyle,rght– zmienne losowe,ghtFit– estymacja parametrów.uogolniony rozkład lambda (Generalized Lambda Distribution) gdzie:
dgld– funkcja gęstości,pgld– dystrybuanta,qgld– kwantyle,rgld– zmienne losowe,gldFit– estymacja parametrów.
Z kolei w bibliotece fGarch są zaimplementowane następujące rozkłady:
uogólniony rozkład błędów (Generalized Error Distribution – GED) gdzie:
dged– symetryczna funkcja gęstości,gedFit– estymacja parametrów,dsged– skośna funkcja gęstości,sgedFit– estymacja parametrów.rozkład t-Studenta (Student-t Distribution) gdzie:
dstd– symetryczna funkcja gęstości,stdFit– estymacja parametrów,dsstd– skośna funkcja gęstości,sstdFit– estymacja parametrów.rozkład normalny (Normal Distribution) gdzie:
dnorm– symetryczna funkcja gęstości,normFit– estymacja parametrów,dsnorm– skośna funkcja gęstości,snormFit– estymacja parametrów.