Jak analizować czas działania kodu R?
Skrypty R działają za wolno?
To z pewnością wina tych leniwych twórców R, którzy coś źle zrobili!
...
A może nie?
A może to nasz kod jest nieefektywnie napisany?
Jeżeli dopuszczasz taką możliwość to przydadzą Ci się narzędzia do analizy czasu działania kodu R.
Czas działania zbioru instrukcji
Funkcja system.time() zlicza jak długo (w sekundach) wykonuje się określony zbiór instrukcji.
Raportowane są trzy liczby, elapsed - czyli ile czasu minęło, user - ile czasu procesora zużył R, system - ile czasu procesora zużyły w tym czasie procesy poza R.
Stwórzmy dużą ramkę danych i zobaczmy jak długo trwa policzenie na niej regresji liniowej.
system.time({
N <- 2000
df <- as.data.frame(matrix(runif(2*N*N), 2*N, N))
tmp <- summary(lm(V1~., data=df))
})
## user system elapsed
## 14.472 0.280 14.794
Kilkanaście sekund!
Profiler
Mierzenie całkowitego czasu działania może być przydatne, choć często chcielibyśmy czegoś więcej. Wiadomo, że funkcja lm() wywołuje pod funkcje. Która z nich zabiera lwią część czasu działania?
Aby to sprawdzić można wykorzystać funkcję Rprof().
Uaktywnia ona profiler, który do określony czas (argument interval, domyślnie 0.02 sek) sprawdza jak wygląda stos wywołań R. Informacje o tym stosie wywołań są zapisywane w pliku wskazanym przez argument filename, domyślnie w pliku Rprof.out.
Rprof("profiler.out", interval = 0.01, memory.profiling=TRUE)
N <- 2000
df <- as.data.frame(matrix(runif(2*N*N), 2*N, N))
tmp <- summary(lm(V1~., data=df))
Rprof()
Fragment pliku Rprof.out jest przedstawiony poniżej. Pierwsze kolumny opisują zajętość pamięci, ostatnia wymienia funkcje, które aktualnie znajdują się na stosie wywołania.
memory profiling: sample.interval=10000
:2707201:50881097:139412560:121:"runif" "matrix" "as.data.frame"
:2707201:50881097:139412560:0:"runif" "matrix" "as.data.frame"
:2707201:50881097:139412560:0:"runif" "matrix" "as.data.frame"
:2707201:50881097:139412560:0:"runif" "matrix" "as.data.frame"
:2707206:58881410:139413624:0:"matrix" "as.data.frame"
:2707206:58881410:139413624:0:"matrix" "as.data.frame"
:2707206:58881410:139413624:0:"matrix" "as.data.frame"
:2707206:58881410:139413624:0:"matrix" "as.data.frame"
:2708210:64700854:140252056:0:"as.vector" "as.data.frame.matrix" "as.data.frame"
Analiza bezpośrednia pliku Rprof.out nie jest łatwa, ponieważ ten plik trzeba odpowiednio przetworzyć aby wyliczyć, która funkcja jak długo działania.
To przetworzenie jest wykonywane funkcją summaryRprof(). Zwraca ona listę z czterema elementami:
by.selfinformacja, która funkcja jak długo działała, uporządkowana zgodnie z czasem działania wyłącznie danej funkcji bez podfunkcji.by.totalinformacja, która funkcja jak długo działała wliczając czas działania podfunkcji,sample.interval, częstość próbkowania kodu,sampling.time, czas trwania całego analizowanego kodu.
Poniżej przedstawiamy przykład działania
summaryRprof("profiler.out")$by.self
## self.time self.pct total.time total.pct
## "lm.fit" 9.84 75.17 9.84 75.17
## "chol2inv" 1.89 14.44 1.89 14.44
## "runif" 0.30 2.29 0.30 2.29
## "na.omit.data.frame" 0.15 1.15 0.26 1.99
## "makepredictcall.default" 0.12 0.92 0.12 0.92
## ".External2" 0.11 0.84 0.42 3.21
## "matrix" 0.08 0.61 0.38 2.90
## "[.data.frame" 0.08 0.61 0.08 0.61
## "as.vector" 0.08 0.61 0.08 0.61
## ".External" 0.07 0.53 0.07 0.53
## "na.omit" 0.05 0.38 0.31 2.37
## "<Anonymous>" 0.03 0.23 13.09 100.00
## "paste" 0.03 0.23 0.10 0.76
## "is.na" 0.03 0.23 0.03 0.23
## "summary.lm" 0.02 0.15 1.91 14.59
## "model.frame.default" 0.02 0.15 0.61 4.66
## "match" 0.02 0.15 0.06 0.46
## "sys.call" 0.02 0.15 0.02 0.15
## "eval" 0.01 0.08 13.09 100.00
## "model.matrix" 0.01 0.08 0.22 1.68
## "model.matrix.default" 0.01 0.08 0.21 1.60
## "makepredictcall" 0.01 0.08 0.13 0.99
## "[[.data.frame" 0.01 0.08 0.09 0.69
## "deparse" 0.01 0.08 0.07 0.53
## "%in%" 0.01 0.08 0.05 0.38
## ".deparseOpts" 0.01 0.08 0.04 0.31
## ".subset2" 0.01 0.08 0.01 0.08
## "all" 0.01 0.08 0.01 0.08
## "c" 0.01 0.08 0.01 0.08
## "is.ordered" 0.01 0.08 0.01 0.08
## "nargs" 0.01 0.08 0.01 0.08
## "paste0" 0.01 0.08 0.01 0.08
## "sum" 0.01 0.08 0.01 0.08
Graficzne statystyki z pakietem profr
Funkcja summaryRprof() przedstawia statystyki działania w sposób tekstowy. Ale często dobrym uzupełnieniem takiej informacji jest wykres, pokazujący jak długo trwają które funkcje i podfunkcje.
Taką graficzną analizę kodu można wykonać pakietem profr (nie zgadniecie kto jest twórcą tego pakietu, tak Hadley Wickham).
Pakiet ten wykorzystać można na dwa sposoby. Można funkcją parse_rprof() odczytać plik utworzony przez Rprof(). Lub (co jest zalecane) profilowanie można wykonać funkcją profr(), która zawiera dodatkowe informacje o czasie wykonania.
Poniżej te dodatkowe informacje są wypisywane, dodatkowo generyczna funkcja plot() przedstawia graficznie stos wywołań funkcji.
library(profr)
pro <- profr({
N <- 2000
df <- as.data.frame(matrix(runif(2*N*N), 2*N, N))
tmp <- summary(glm(V1~., data=df))
}, 0.01)
head(pro)
## level g_id t_id f start end n leaf time source
## 28 1 1 1 as.data.frame 0.00 0.44 1 FALSE 0.44 base
## 29 1 2 1 summary 0.44 23.40 1 FALSE 22.96 base
## 30 2 1 1 matrix 0.00 0.33 1 FALSE 0.33 base
## 31 2 2 1 as.data.frame.matrix 0.33 0.44 1 FALSE 0.11 base
## 32 2 3 1 glm 0.44 21.20 1 FALSE 20.76 stats
## 33 2 4 1 summary.glm 21.20 23.40 1 FALSE 2.20 stats
plot(pro)
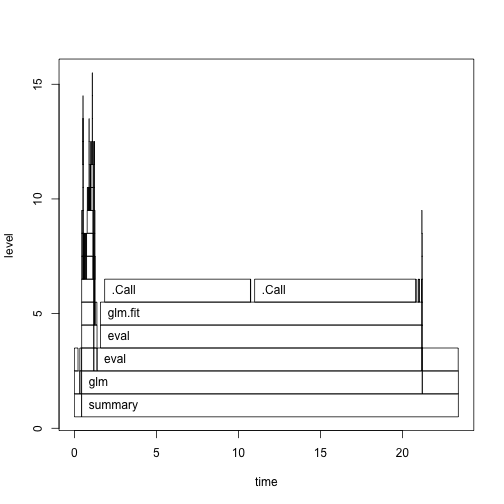
Na bazie tego jednego wykresu łatwo zauważyć, że generowanie danych trwało chwilę, wywołanie summary() trwało trochę dłużej, a najwięcej czasu zabrała funkcja glm()
Ale po co nam to?
Mierząc czas działania, możemy porównać kilka potencjalnych rozwiązań i wybrać lepsze.
Zobaczmy na przykład, w jaki sposób tworzyć wektor 100 000 losowych liczb z rozkładu jednostajnego. Porównamy cztery możliwe rozwiązania i zobaczymy które jest najszybsze.
system.time({ x=NULL; for (i in 1:10^5) x =c(x, runif(1)) })
## user system elapsed
## 17.573 13.793 31.471
system.time({ x=NULL; for (i in 1:10^5) x[i] =runif(1) })
## user system elapsed
## 10.191 13.802 24.573
system.time({x=numeric(10^5); for(i in 1:10^5) x[i]=runif(1)})
## user system elapsed
## 0.375 0.059 0.482
system.time({ x=NULL; x = runif(10^5) })
## user system elapsed
## 0.004 0.000 0.003
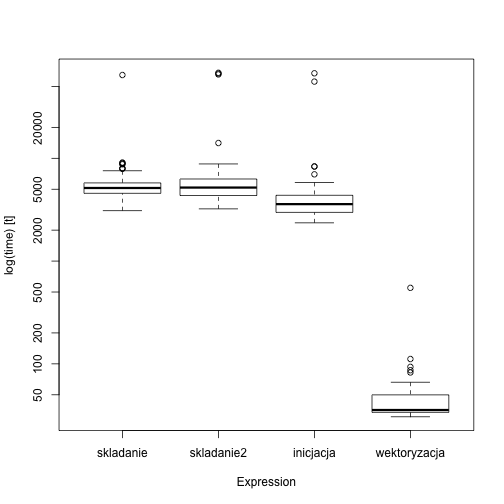
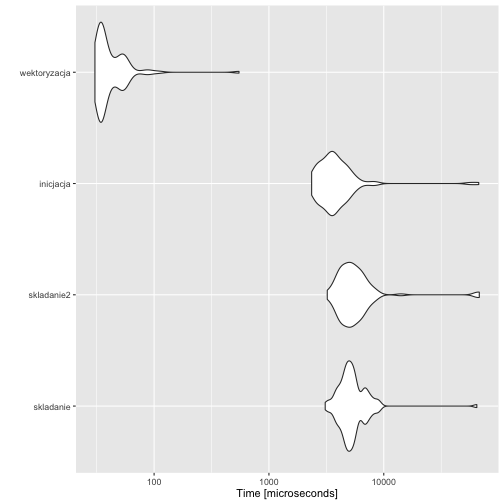
Pierwsze dwa dynamicznie rozciągają wektor, co w R oznacza kopiowanie w kółko całego wektora wyników. Trwa to długo. Inicjacja wektora pustymi wartościami powoduje że czas obliczeń skrócił się 30 razy. Wektoryzacja obliczeń skraca czas obliczeń o kolejne 10 razy.
Z dokładnością do mikrosekundy
Jeżeli mierzenie czasu działania z dokładnością do sekundy nie jest wystarczające, to warto skorzystać z biblioteki microbenchmark. Nie dość, że powtarza ona określone instrukcje wiele razy (domyślnie 100), to jeszcze prezentuje graficznie i tekstowo rozkład czasów działania.
W poniższym przykładzie porónujemy ponownie cztery sposoby losowania wektora liczb.
library(microbenchmark)
library(ggplot2)
pro <- microbenchmark(
skladanie = { x=NULL; for (i in 1:10^3) x =c(x, runif(1)) },
skladanie2 = { x=NULL; for (i in 1:10^3) x[i] =runif(1) },
inicjacja = {x=numeric(10^3); for(i in 1:10^3) x[i]=runif(1)},
wektoryzacja = { x=NULL; x = runif(10^3) })
boxplot(pro)
autoplot(pro)
Materiały
Kilka bardzo ciekawych zadań do rozwiązania można znaleźć na stronie: